
__________________________________________
3.7.- Cuando se suplementa con los compuestos A, B, C, D, E, F, G, H e I el medio mínimo en el que se cultivan las cepas mutantes nutricionales de Neurospora 1 al 9 se obtienen los siguientes resultados:

Publicado el 20 septiembre 2008 - 08:19


Publicado el 24 septiembre 2008 - 03:21






Publicado el 28 septiembre 2008 - 10:27


Publicado el 29 septiembre 2008 - 07:44
Publicado el 02 octubre 2008 - 04:14

Publicado el 04 octubre 2008 - 03:13

Publicado el 04 octubre 2008 - 07:00
Publicado el 05 octubre 2008 - 07:06

Publicado el 07 octubre 2008 - 10:18

Publicado el 11 octubre 2008 - 01:16


Publicado el 14 octubre 2008 - 06:15

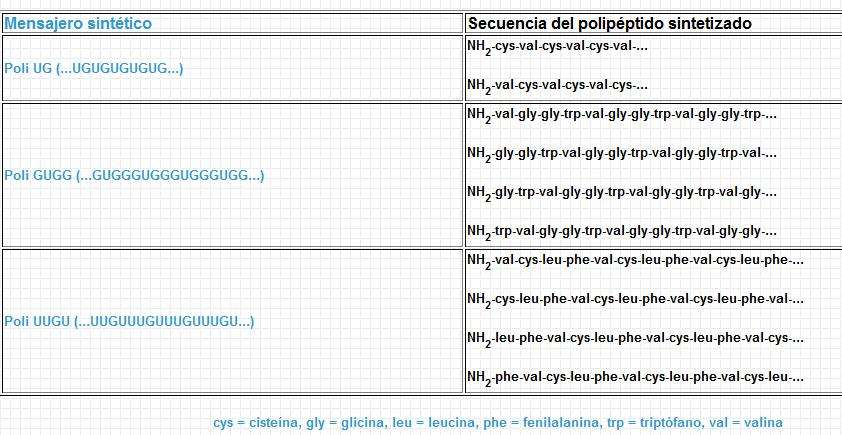
Publicado el 16 octubre 2008 - 01:05



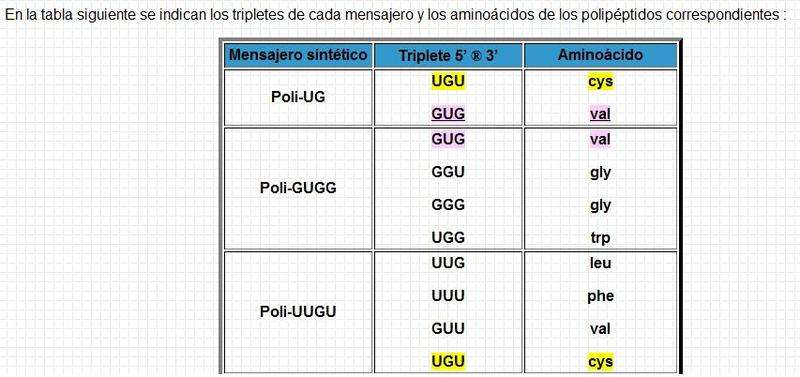

Publicado el 19 octubre 2008 - 08:19

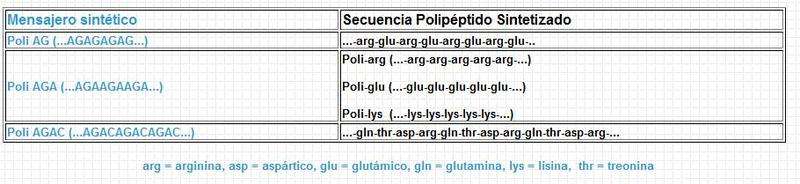
Publicado el 22 octubre 2008 - 06:17


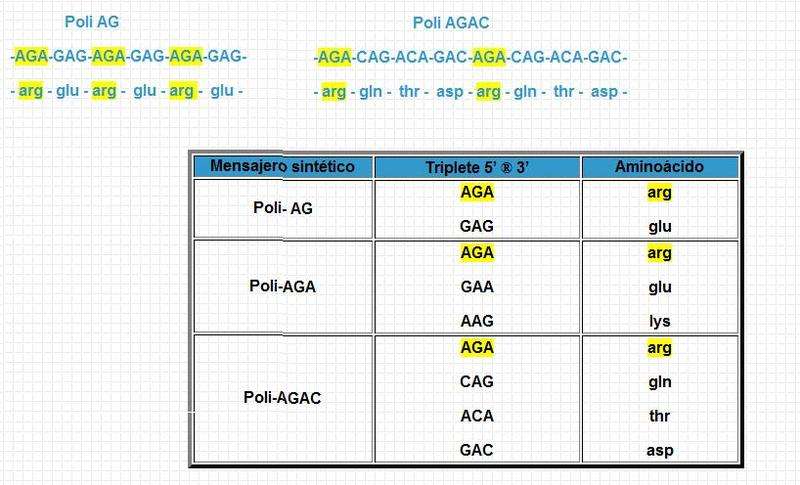

Publicado el 27 octubre 2008 - 03:45

Publicado el 30 octubre 2008 - 11:31


Publicado el 03 noviembre 2008 - 05:58

Publicado el 11 noviembre 2008 - 04:36


Publicado el 12 noviembre 2008 - 05:29

Publicado el 13 noviembre 2008 - 12:19

